首个家山羊参考基因组解析与技术创新
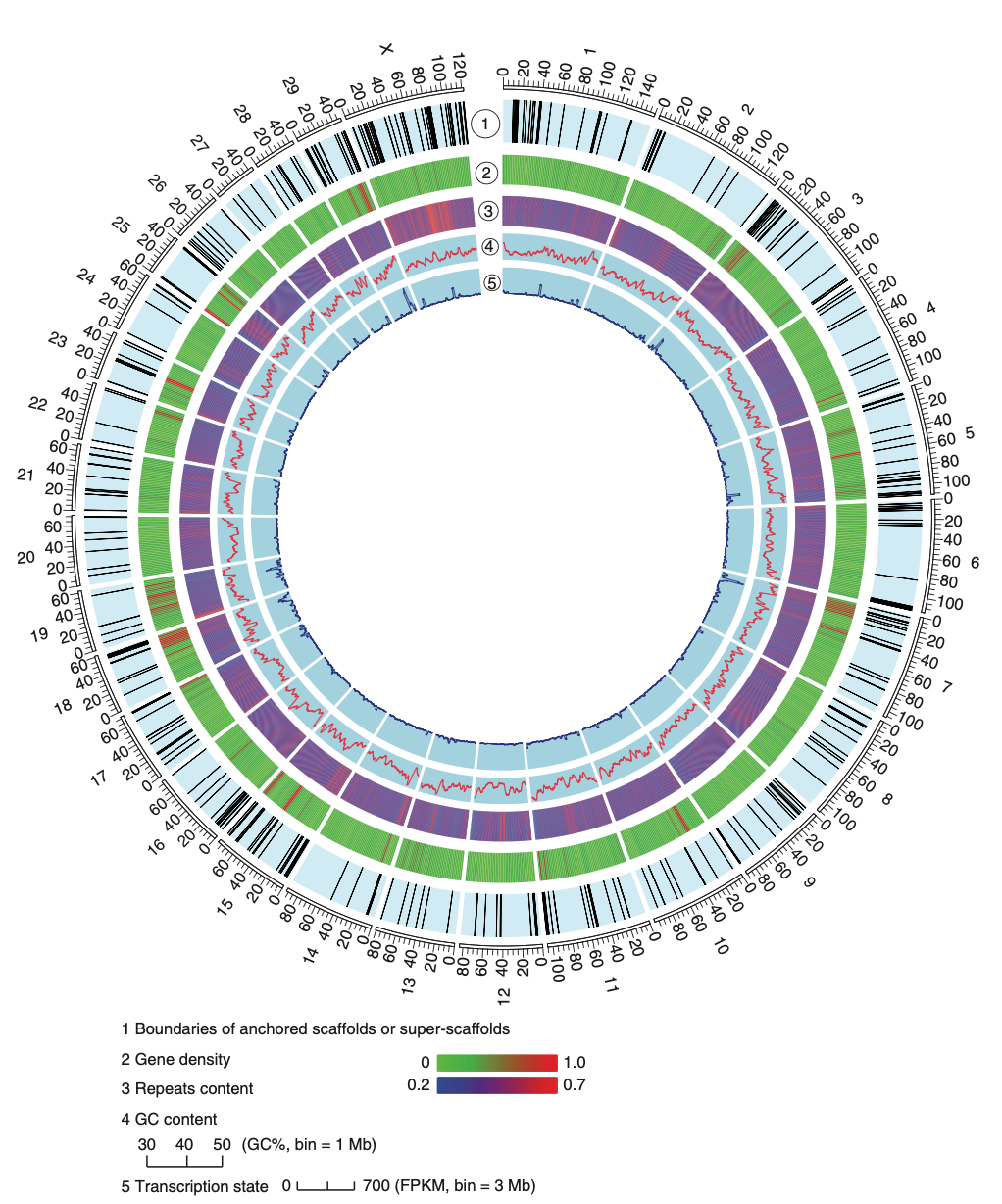
我们参与的研究发表于《Nature Biotechnology》,通过引入全基因组光学图谱技术,成功构建了高质量的云南黑山羊参考基因组。
由 XinLab 团队成员参与,与中国科学院昆明动物研究所、华大基因等机构合作完成的研究论文 “Sequencing and automated whole-genome optical mapping of the genome of a domestic goat (Capra hircus)” 已在 Nature Biotechnology 发表。该研究通过技术集成,解决了大型反刍动物基因组组装的瓶颈。
研究背景
山羊是最早被驯化的家畜之一,也是全球重要的肉、乳、纤维来源。然而,在 2013 年之前,山羊基因组的缺失严重制约了对其优良性状遗传基础的解析。由于大型哺乳动物基因组含有大量重复序列,仅靠短读长测序很难获得高连续性的组装结果。
核心突破
-
光学图谱(Optical Mapping)技术的成功应用 研究团队首次在大规模基因组组装中引入了高通量全基因组光学图谱技术。通过这一技术,组装的超支架(Super-scaffolds)N50 达到了 16.3 Mb,比传统方法提升了 5 倍以上。这证明了光学图谱在辅助染色体水平组装中的巨大潜力。
-
基因组特征解析 研究获得了约 2.66 Gb 的山羊基因组序列,预测了 22,175 个蛋白质编码基因。通过比较基因组学分析,研究揭示了反刍动物独特的代谢通路以及与免疫、产乳相关的基因演化。
-
山羊与绵羊的差异演化
研究通过对山羊、绵羊及其他牛科动物的选择压力分析,定位了导致山羊在环境适应性(如耐粗饲、抗病性)方面优于其他反刍动物的特定遗传变异。
研究意义
这一高质量参考基因组的发布,为全球山羊遗传学研究提供了一个“坐标系”。它极大地推动了山羊抗病育种、毛皮品质改良以及繁殖效率提升的分子标记辅助育种进程。
协作与致谢
本项目由中科院昆明动物研究所王文研究员团队、华大基因(BGI)及多家国际科研机构共同完成。XinLab 团队(刘心、谢敏等)在基因组测序、光学图谱数据整合及生物信息分析中发挥了重要作用。